即可将网页分享至朋友圈
近日,电子科技大学生命科学与技术学院真菌与植物功能基因组学研究团队在国际知名期刊Nature Communications发表了题为“High performance TadA-8e derived cytosine and dual base editors with undetectable off-target effects in plants”的研究论文。该研究在水稻和番茄中对一系列TadA-8e来源的工程化胞嘧啶和双碱基编辑系统进行比较,筛选出具有高编辑活性和较窄编辑窗口的TadCBEa、TadCBEd和TadCBEd_V106W作为高效的CBE,以及一个单一脱氨酶结构域同时实现C-to-T和A-to-G的双碱基编辑系统TadDE。研究展示了TadCBEa和TadDE在水稻中进行多位点碱基编辑时具有高编辑活性和高编辑特异性,并列举了TadDE在作物功能获得性的工程化改造中的应用,极大地丰富了植物基因组编辑工具箱。

本研究开发并展示了TadA-8e来源的胞嘧啶碱基编辑系统TadCBEs和双碱基编辑系统TadDE能够用于高效率、高纯度、高特异性的植物基因组编辑,为水稻等农作物的种质创新带来更多的可能性,具有巨大的应用潜力。全文主要研究结果概述如下:
1、在水稻细胞中比较不同碱基编辑系统的编辑活性
为了研究TadA-8e来源的CBEs(TadCBEs)是否能够在植物中有效地实现C-to-T,构建了一系列适用于植物系统的TadCBEs,包括TadCBEa、TadCBEd、TadCBEd_V106W、eTd-CBE和Td-CBEmax,以及双碱基编辑系统TadDE和TadDE-NG(图1a),在20个水稻内源位点处进行编辑活性的评价。结果表明,大多数TadCBEs和TadDE在水稻细胞中实现有效的C-to-T碱基编辑(图1b,c)。在20个目标位点中,TadDE在16个位点检测到C-to-T和A-to-G的同时编辑,且A-to-G碱基编辑效率与ABE8e相当(图1b,d)。所有这些碱基编辑系统的插入和缺失(indel)效率都非常低(图1b,e)。
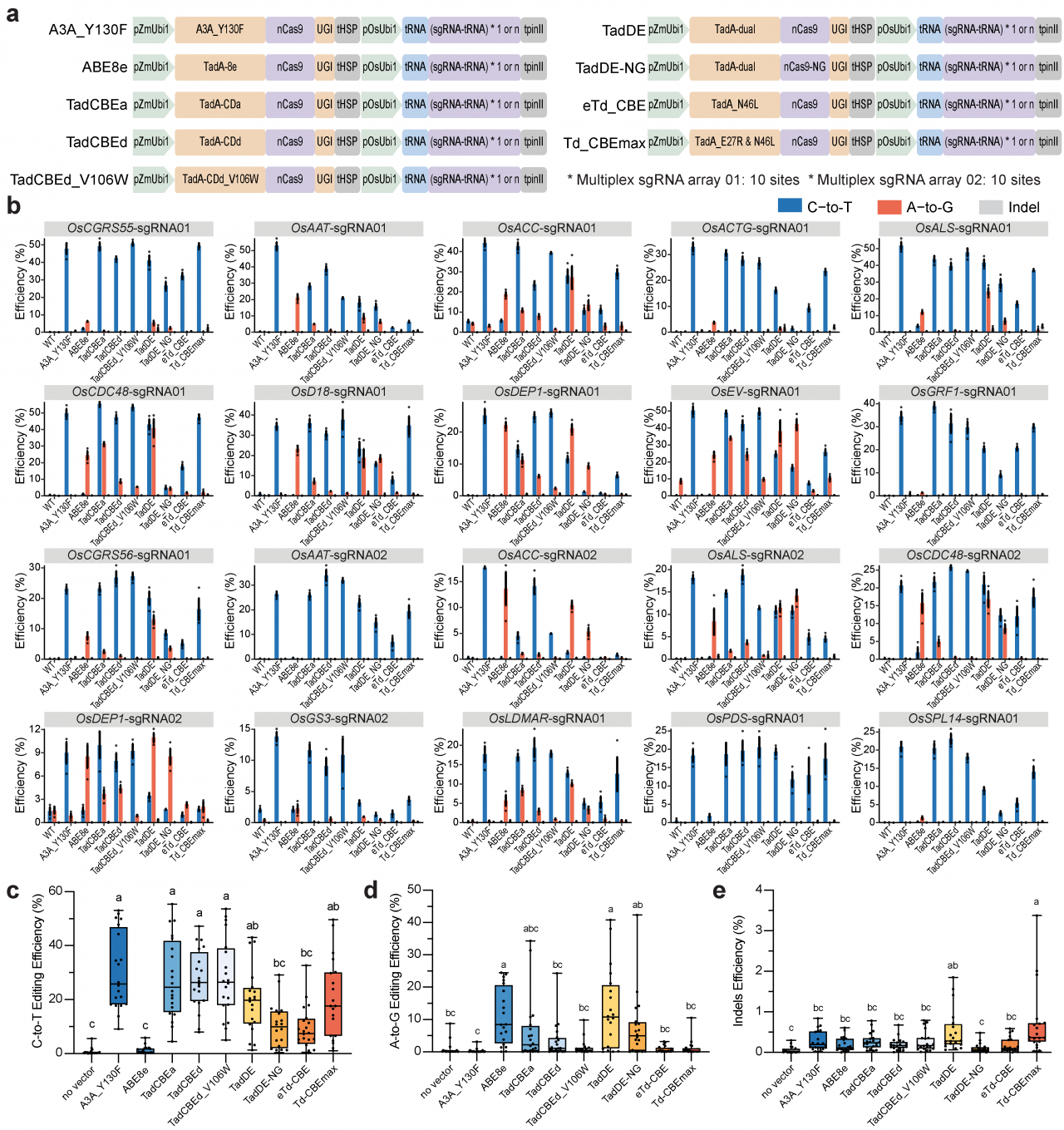
图1:TadA-8e来源的碱基编辑系统在水稻细胞中的编辑活性评价
2、TadA-8e来源的CBEs和双碱基编辑系统在水稻细胞中的编辑特征
对20个靶向位点处的碱基编辑结果进行表征,结果表明,与A3A_Y130F的C-to-T编辑窗口跨越protospacer的第3~16位相比,TadCBEa、TadCBEd和TadCBEd_V106W的C-to-T编辑窗口较窄,集中在protospacer的第4~8位,这与ABE8e的A-to-G碱基编辑窗口一致(图2a)。此外,TadDE的C-to-T碱基编辑窗口和A-to-G碱基编辑窗口几乎重叠(图2a)。这些数据表明,尽管改变了碱基编辑序列偏好,工程化改造后的TadA脱氨酶变体仍保留了TadA-8e的通用碱基编辑窗口。编辑后的序列表明,双碱基编辑系统TadDE在20个目标位点中均实现了C-to-T和A-to-G的同时编辑(图2b)。
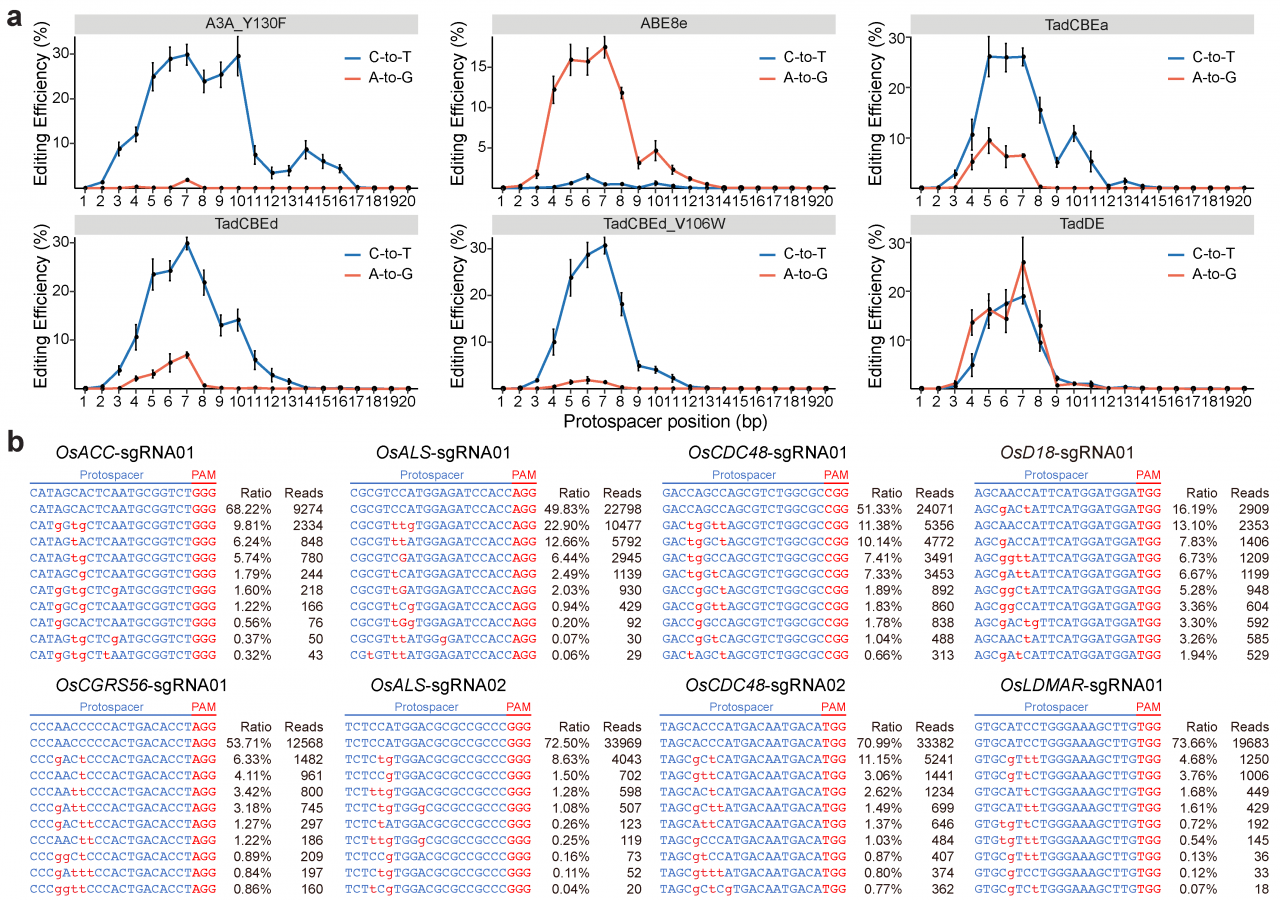
图2:不同碱基编辑系统在水稻细胞中的编辑特性分析
3、TadA-8e来源的碱基编辑系统在番茄细胞中实现有效编辑
为了评估TadA-8e来源的CBEs和双碱基编辑系统在双子叶植物中的有效性,研究者在番茄原生质体中进行了测试。结果表明,TadCBEs和TadDE在四个靶向位点处均有效实现了C-to-T编辑(图3a,b),TadDE显示出有效的A-to-G编辑(1.3%-10.0%)(图3c)。所有TadA-8e来源的碱基编辑系统的插入缺失频率都非常低(图3d)。与水稻原生质体中的数据一样,TadA-8e来源的CBE在番茄细胞中表现出比A3A_Y130F更窄的碱基编辑窗口(图3e),TadDE实现的C-to-T和A-to-G的碱基编辑窗口重叠(图3e),TadDE双碱基编辑系统在SlBlc-sgRNA01和SlBlc-sgRNA02位点的编辑事件表明TadDE在番茄细胞中实现了C-to-T和A-to-G的同时编辑(图3f)。
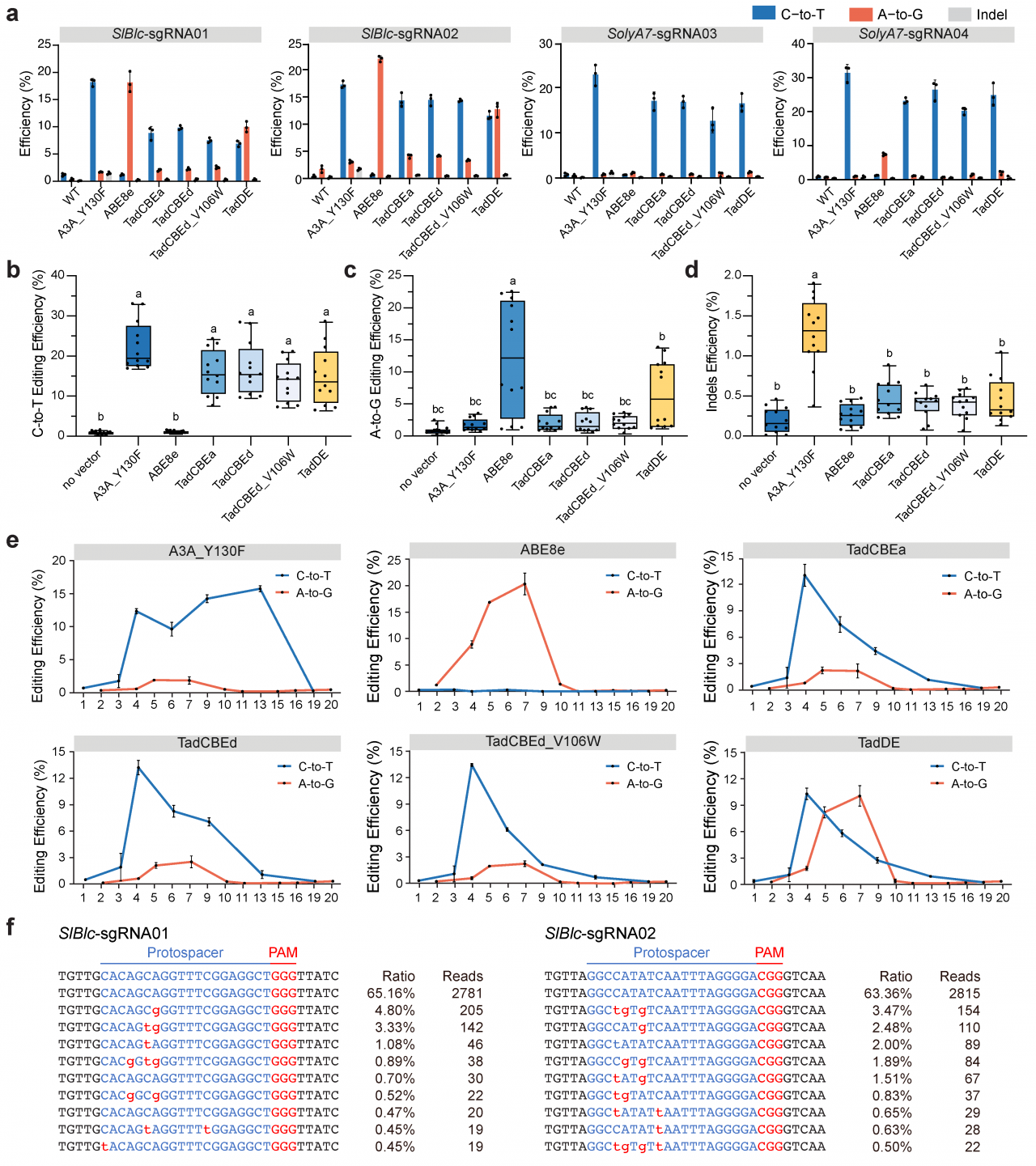
图3:TadCBEs和TadDE碱基编辑系统在番茄细胞中的编辑活性评价
4、TadCBEa和TadDE在水稻植株中实现了有效的单位点或多位点编辑
接下来评估TadCBEa和TadDE在水稻植株中的碱基编辑活性(图4a),对转基因再生苗进行全基因组测序(WGS)(图4b)。分析结果表明,TadCBEa和TadDE在水稻植株中能够实现有效的C-to-T碱基编辑(图4c),且大多数位点处实现了高比例的双等位基因编辑(图4c)。由于TadDE编辑材料中几乎未检测到A-to-G碱基编辑效率(图4d),为了明确TadDE在水稻植株中的有效性,使用TadDE对6个位点进行单位点碱基编辑(图4e),转基因T0代植株的基因型鉴定结果显示,在6个目标位点中,有五个目标位点实现了高效的C-to-T和A-to-G同时编辑(图4f、4g)。

图4:TadCBEa和TadDE碱基编辑系统在水稻植株中的编辑活性评价
5、TadCBEa和TadDE在水稻植株中的编辑特异性分析
对TadCBEa和TadDE多位点编辑水稻材料进行全基因组测序以明确编辑器在基因组范围内的脱靶编辑(图5a),结果表明,TadCBEa和TadDE都未检测到基因组范围内的脱靶编辑(图5b-h)。对TadCBEa和TadDE在转录组水平上的脱靶效应进行了分析(图5a),与组织对照相比,TadCBEa和TadDE碱基编辑系统没有在水稻转录组中引入额外的C-to-U和A-to-I突变(图5i-m)。TadCBEa和TadDE在DNA和RNA水平上均未引发显著的脱靶编辑,表明它们在植物中是高度特异的基因组编辑工具。
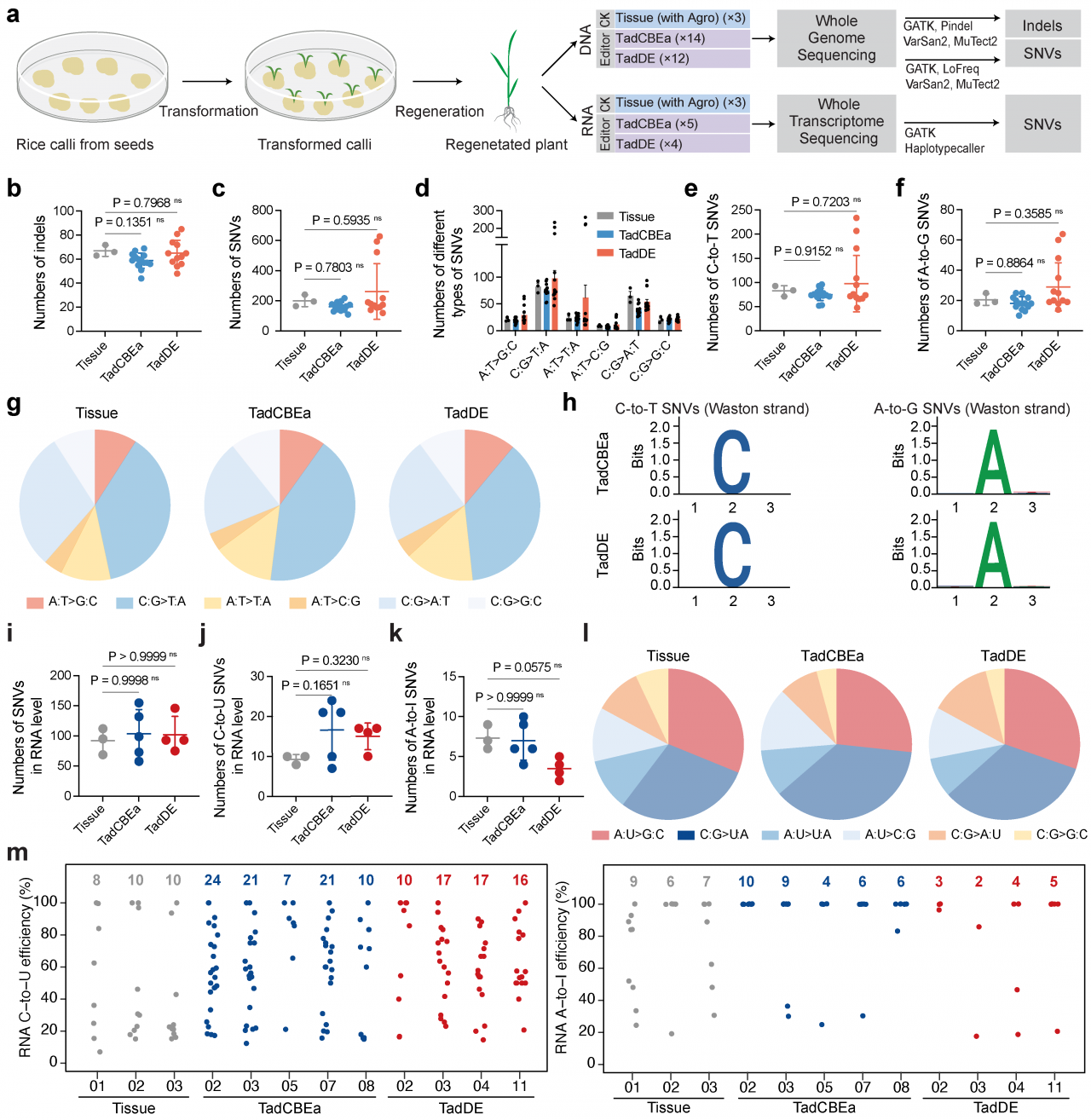
图5:TadCBEa和TadDE碱基编辑系统在水稻植株中的编辑特异性分析
6、TadDE双碱基编辑系统赋予水稻除草剂抗性
TadDE编辑系统编辑OsALS位点以对水稻的除草剂抗性进行工程化改造,获得了一个sgR17突变的除草剂抗性水稻单株(B1-01),以及五个sgR18突变的除草剂抗性水稻植株(B2-04至B2-08)(图6a-c)。B1-01单株携带了一个R190H突变(图6d),而B2-04至B2-08单株携带了G628E和G629S的单突或双突(图6e)。结构分析显示,这些突变都影响了OsALS酶的催化核心(图6f),阻止了草甘膦对水稻生长的抑制作用。TadDE在水稻中进行功能获得性的工程化改造,赋予了植株除草剂抗性。
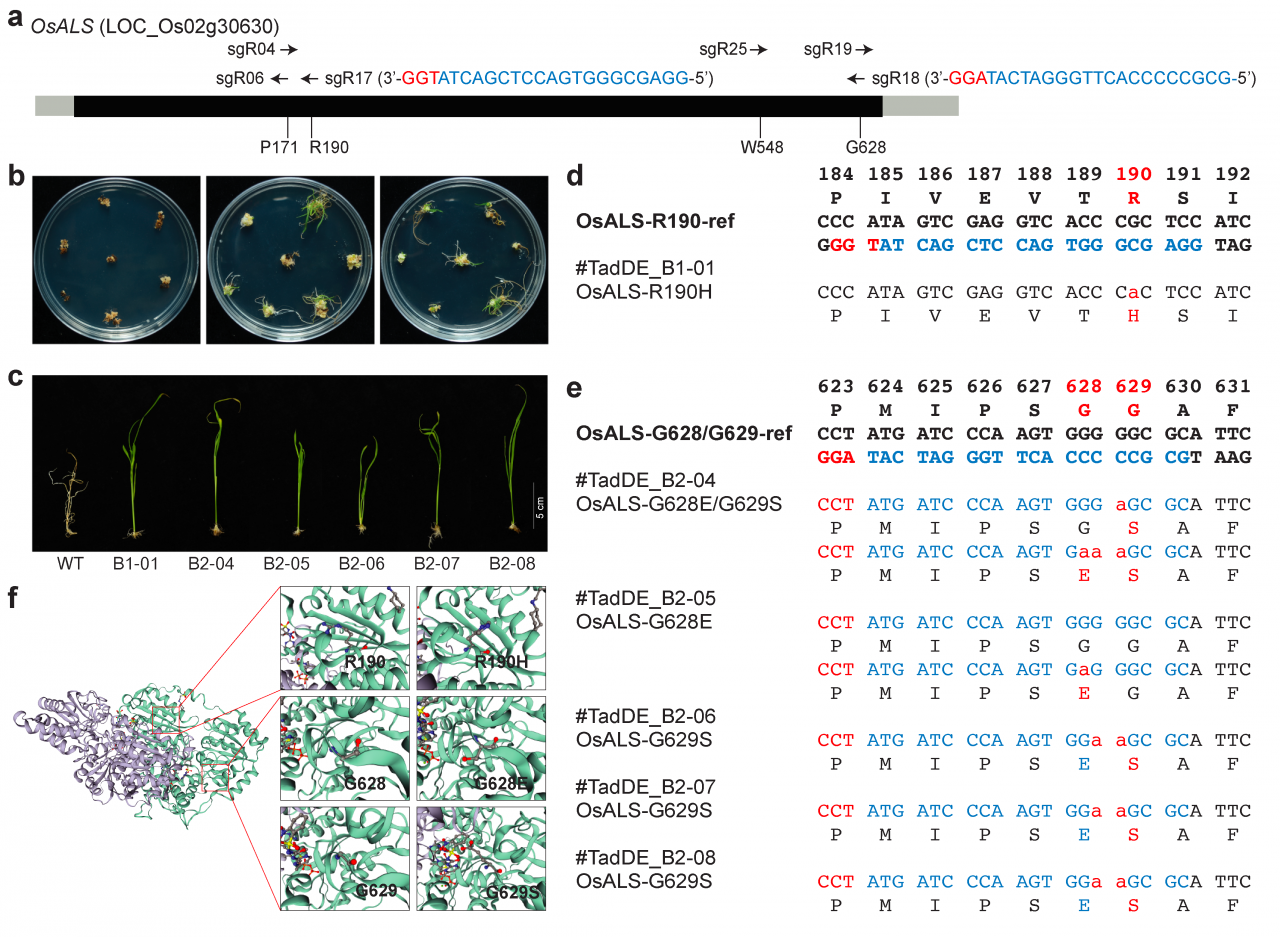
图6:TadDE双碱基编辑系统在工程化改造水稻除草剂抗性中的应用
7、TadDE调控miRNA对OsSPL14的抑制作用以提升OsSPL14基因表达水平
应用TadDE双碱基编辑系统对OsSPL14基因序列中的miRNA156靶位点进行碱基编辑(图7a)。在野生型植物中,miRNA156抑制了OsSPL14的转录;理想情况下,如果能在OsSPL14的miRNA156靶位点引入同义突变,那么所得到的编辑后的OsSPL14仍将编码相同的蛋白质,但其转录本将对miRNA156介导的靶向剪切具有抗性,因此预期在编辑植株中会有更多的OsSPL14转录本和蛋白表达(图7b)。在再生的T0代水稻苗中,我们鉴定出了六个编辑植株(图7c),其中TadDE_B4-16和TadDE_B4-18两个单株各携带了一个在miR156靶位点的同义突变,这是由TadDE对非编码链进行的A-to-G编辑引起的(图7c)。qPCR分析显示,与野生型相比,两个编辑植株中的OsSPL14表达量显著上调(图7d),此应用展示了TadDE通过突变miRNA靶位点而未改变编码蛋白质序列来上调靶基因的应用。
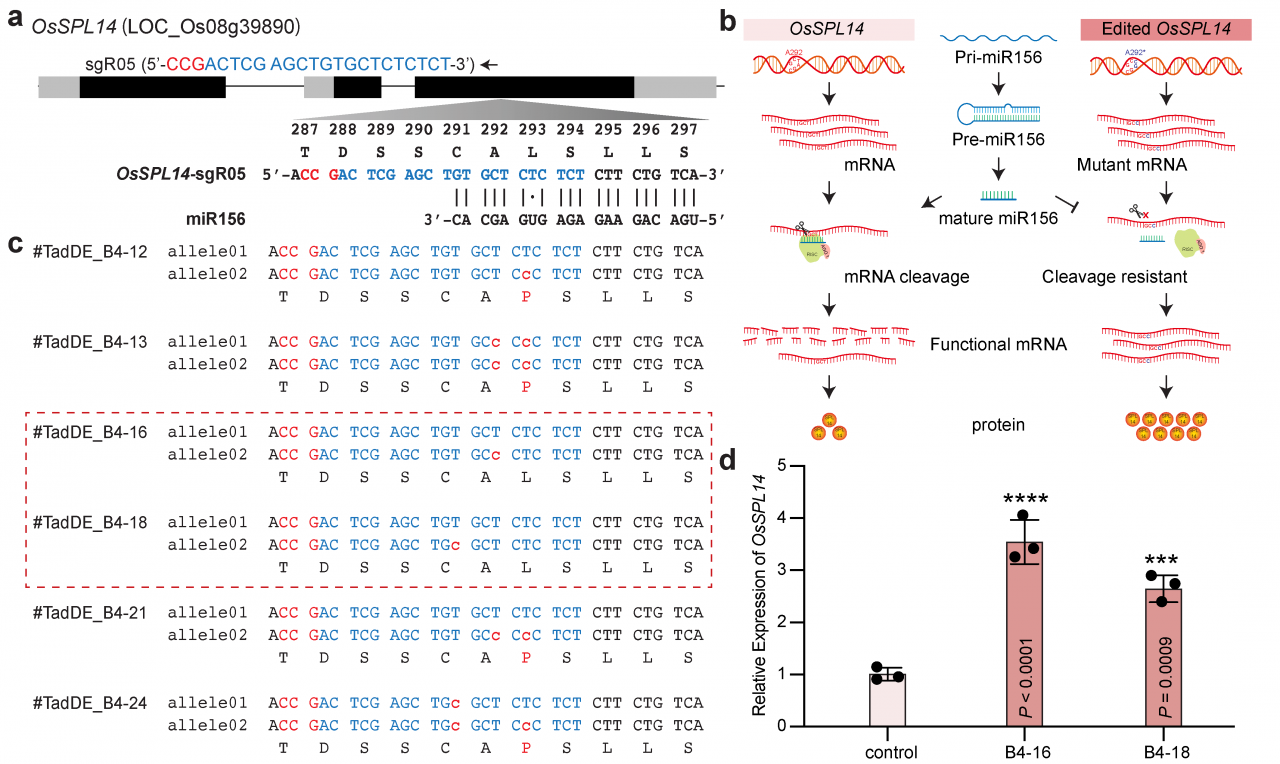
图7:TadDE双碱基编辑系统在调控miRNA剪切OsSPL14中的应用
生命科学与技术学院2021级博士生范婷婷、2019级博士生刘诗诗,马里兰大学Yanhao Cheng博士,扬州大学博士生吴越超、西南大学唐旭博士为论文共同第一作者,电子科技大学张勇教授、马里兰大学Yiping Qi教授及扬州大学张韬教授为论文共同通讯作者,研究工作得到了国家科技重大专项、国家自然科学基金、四川省创新人才等项目的资助。
论文链接:https://www.nature.com/articles/s41467-024-49473-w
编辑:刘瑶 / 审核:李果 / 发布:陈伟


